Neurologie
Sclérose en plaques : causes ancestrales de la prédisposition génétique des européens
Pourquoi les européens sont-ils plus susceptibles à la sclérose en plaque ? Une étude de génétique des populations fait la lumière sur les origines ancestrales de la prédisposition génétique. L’histoire des migrations ne serait pas sans effets.
- NLshop/istock
Les maladies auto-immunes comme la sclérose en plaques (SEP) sont comprises comme la résultante d’une interaction entre une susceptibilité génétique, un environnement et un mode de vie. Du côté environnemental, le virus Epstein-Barr a été soulevé récemment comme un élément causal de l’apparition de la maladie (Bjornevik et al., 2022). Du côté génétique, le risque est polygénique et concerne essentiellement les populations européennes et nord-africaines.
Cette sélectivité de population soulève une question anthropologique (Froment, 2013): celle des causes « distales » de la SEP, c’est-à-dire l’origine de sa susceptibilité dans l’histoire évolutive de l’espèce humaine, qu’on oppose aux causes « proximales », à l’échelle de la vie d’un individu et auxquelles on s’intéresse habituellement en médecine. Une étude de génétique des populations est paru ce mois-ci dans Nature et mets en cause un épisode de grande migration il y a 5000 ans (Barrie et al., 2024).
Forte prévalence en Europe et gradient Nord-Sud
L’incidence de la SEP est en hausse sur les 50 dernières années et est très hétérogène selon les populations et les régions géographiques. La prévalence la plus forte est en Europe du Nord. Le gradient nord-sud de l’incidence de la SEP est généralement attribué à des facteurs environnementaux tel que l’insuffisance en vitamine D. L’auto-immunité peut également résulter d’une pression de sélection par des pathogènes au cours de l’histoire évolutive de l’espèce humaine, qui aurait déséquilibré les voies de signalisation pro- et anti-inflammatoires. A ce titre, cette étude montre que ce gradient corrèle avec différents héritages génétiques de populations anciennes, ce qui indique une explication plutôt génétique en lien avec l’histoire des migrations et métissages sur le continent européen.
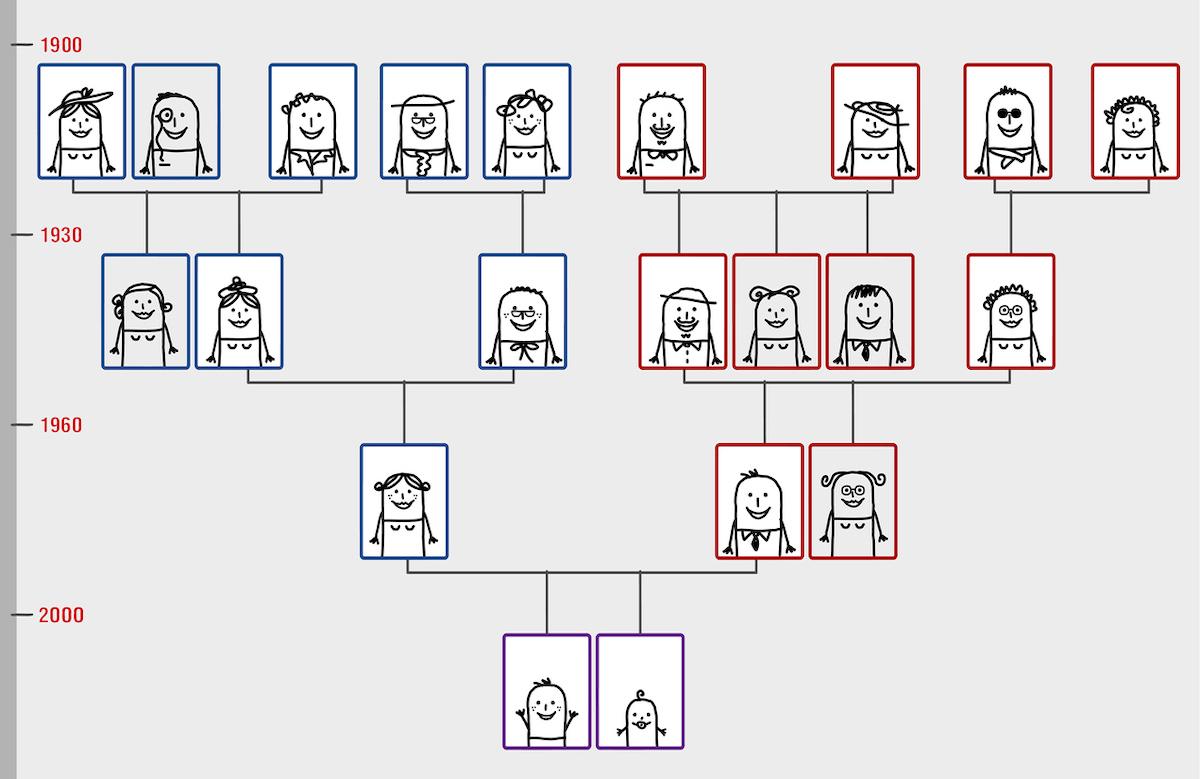
Le mode de vie originel de l’espèce humaine est celui de « chasseur-cueilleur nomade ». Sur les 10 000 dernières années, il y a eu deux changements majeurs de mode de vie : l’agriculture et l’élevage, qui ont émergés séparément dans des populations aux fonds génétiques différents. Les chasseurs-cueilleurs occidentaux sont la population de base du continent européen apparues il y a 24 000 ans (fin de la dernière glaciation). Elle fut largement remplacée durant le néolithique (apparition de l’agriculture ; -7000 à -3000 ans) par des agriculteurs issus d’Anatolie, poussés par la densification de la population. C’est ladite « transition néolithique ». L’ancestralité des steppes apparaît plus tard autour de la mer Caspienne par le métissage des chasseurs-cueilleurs orientaux (actuelle Russie Occidentale) et caucasiens (actuel Iran). Cette population, au mode de vie pastoral, a fait une migration massive de la steppe eurasienne vers l’Europe durant l’âge de Bronze (métallurgie ; -3000 à -800 ans), également poussée par la densification de la population, au point de rendre compte de la moitié patrimoine génétique européen.
Les études d’associations pangénomiques (en anglais : genome-wide association studies ; GWAS) génotypent les individus uniquement selon des puces de variants ponctuels (en anglais : single nucléotide polymorphism ; SNP) plutôt que par séquençage. Les allèles sont imputés à partir des patterns de SNP couvrant les séquences de leurs gènes grâce à des bases de données de référence (ici : 1000 génomes). Les GWAS permettent d’évaluer « l’ancestralité » des individus, c’est-à-dire la part de patrimoine génétique héritée d’une population particulière. Les GWAS ont également identifié 233 gènes de susceptibilité à la SEP (International Multiple Sclerosis Genetics Consortium, 2019). L’association la plus forte est le portage de l’allèle HLA-DRB1*15:01 avec un risque relatif de 3. Dans l’ensemble, la contribution de ce « risque génétique » de 233 gènes au risque de SEP est estimée à 30%. Le reste est attribué à l’environnement (comme une infection tardive à EBV) et au mode de vie (tabagisme, obésité durant l’adolescence, nutrition). Cette étude analyse l’apparition et l’évolution de la fréquence du risque génétique de la SEP au regard de l’Histoire européenne grâce à des GWAS faits sur des spécimens archéologiques allant de la préhistoire à nos jours.
Une étude de génétique des populations
Les ancestralités vis-à-vis des populations anciennes des génomes européens modernes ont été caractérisés sur 410 000 génotypes de britanniques s’identifiant comme « blancs » issus de la UK Biobank. Pour établir ces ancestralités anciennes et suivre l’évolution des génotypes au cours de l’Histoire, les chercheurs ont utilisé une base de données publiée de 1347 génomes anciens, à laquelle fut ajouté 318 prélèvements archéologiques d’ADN issues d’Eurasie et couvrant une période allant du mésolithique (réchauffement climatique post-glaciation ; -10 000 à -7000 ans) à l’âge de Bronze.
Ces prélèvements viennent majoritairement de dents, puis de parties pétreuses d’os temporaux, puis d’os longs. Une base de données de 86 prélèvements couvrant la période médiévale et postmédiévale au Danemark fut également utilisée. Après séquençage de ces prélèvements, 1750 génomes anciens ont pu être imputés par méthodes bioinformatiques. Les chercheurs ont estimé la part d’ancestralité des populations anciennes dans ces génomes, recherchés des signes de sélection positive des gènes de susceptibilité connus tout en investiguant des associations à des événements environnementaux.
Importance de la proportion d’ancestralité de la population pastorale des steppes
La première apparition du SNP le plus caractéristique du portage HLA-DRB1*15 :01 fut trouvé chez un individu du néolithique en Italie et daté à environ -5700. La fréquence du portage augmenta rapidement aux alentours d’il y a 5300 ans, ce qui correspond à l’émergence de la culture Yamnaya (- 5000 à – 2500 ans) dans la population des steppes et ses dérivées. De nos jours, la fréquence du portage HLADRB1*15 :01 est la plus élevée chez les individus de la UK Biobank dont l’ancestralité moderne correspond aux pays scandinaves et dont l’ancestralité ancienne correspond aux populations des steppes.
L’importance de la proportion d’ancestralité de la population pastorale des steppes corrèle avec le gradient nord-sud d’incidence de la SEP. Les régions du génome avec les variations d’ancestralité les plus extrêmes sont le locus LCT/MCM6 du chromosome 2, impliqué dans la régulation de la persistance de la lactase, et le complexe majeur d’histocompatibilité du chromosome 6, codant les gènes HLA.
Les chercheurs ont calculé un « score de risque ancestral » de SEP qui est le plus élevé pour l’ancestralité des steppes (après les 3 ancestralités de chasseurs-cueilleurs, puis les agriculteurs). Dans la région du HLA, presque tous les SNPs du risque génétique de SEP sont le plus probablement hérités de l’ancestralité des steppes.
Au-delà de l’association de l’ancestralité des steppes au risque de SEP, les chercheurs ont analysé si les SNPs ont été sélectionnés positivement ou négativement en suivant l’évolution des fréquences de SNPs au fil des âges tant en ajustant pour les métissages de population. La fréquence des SNPs associés à la SEP a effectivement augmenté au sein même de l’ancestralité des steppes notamment durant la période de la migration vers l’Europe il y a 5000 ans.
Pour investiguer la nature de cette sélection, les chercheurs ont mesuré des déséquilibres de liaison entre les SNPs vis-à-vis de leurs ancestralités locales. Ceci a permis de scorer dans certaines régions chromosomiques les événements de recombinaison chromosomique impliquant des ancestralités différentes. En référence, la région LCI/MCM6 (régulation de la lactase) a eu peu de recombinaisons d’ancestralité, ce qui est mis sur le compte d’une pression de sélection plus récente (régime incluant des produits laitiers). En revanche, la région du HLA en a eu beaucoup, ce qui indique une sélection d’individus porteurs de régions HLA métissées. La région des HLA de classe 1 était notamment diverse sur un plan ancestral tout en étant fortement contrainte génétiquement.
Outre l’association des SNPs à des maladies, les registres bioinformatiques de la UK Biobank et FinnGen répertorient tous les traits statistiquement associés aux SNPs. Ainsi, les chercheurs ont pu examiner les traits associés aux 32 SNPs qui ont été significativement sélectionnés à cette époque. en dehors de la SEP, qui si elle existait à l’époque, ne devait pas conférer un avantage évolutif. La littérature de ces SNPs les associe à une protection contre des infections virales chroniques (EBV, VZV, HSV, CMV), des infections virales aiguës qui nécessitent des communautés plus grandes qu’un groupe de chasseur-cueilleur pour être transmises (ex : grippe et oreillons), des infections parasitaires, des infections bactériennes respiratoires, cutanées, intestinales ou sexuellement transmissibles. Toutefois, aucune association n’était significative dans les bases de GWAS après l’ajustement pour tests multiples. Les auteurs avancent un manque de puissance pour associer les SNPs à des maladies infectieuses, car elles sont devenues rares à l’ère vaccinale.
En comparaison, la polyarthrite rhumatoïde (PR), une maladie auto-immune systémique dont l’allèle le plus associé est HLA-DRB1*04:01. Les SNPs associés à ces allèles ont continuellement été sélectionnés négativement sur les 15 000 dernières années et sont associés aux ancestralités de chasseurs-cueilleurs occidentaux et orientaux. Eux aussi sont pour la majorité associés à une protection contre des infections. Ces infections recouvrent celles contre lesquelles les SNPs associés à la SEP protègent et en incluent d’autres (ex : amoebose, rougeole, hépatites, arboviroses,…).
Le rôle de la coévolution des infections et des hommes
La coévolution de pathogènes avec leurs hôtes humains dans des populations séparées et aux modes de vie différents a entrainé une sélection d’allèles de gènes impliqués dans les réponses immunitaire propre à chaque ancestralité. Cette étude montre que le mélange des populations a favorisé la sélection de régions HLA recombinées, incluant un fort héritage de l’ancestralité des steppes. Le néolithique tardif et l’âge de Bronze furent marqués par une forte hausse des maladies infectieuses, ce qui est en lien avec une densification des populations et un contact augmenté avec les animaux domestiqués et leurs produits. Les spécimens archéologiques ne permettent pas d’identifier les maladies précises de cette époque. Toutefois, on sait que le dernier ancêtre commun à de nombreux pathogènes modernes remonte à cette époque.
Notre époque est aussi marquée par un changement de mode de vie et d’environnement, notamment vis-à-vis de l’hygiène, ce qui ne favorise plus l’architecture génétique sélectionnée à l’âge de Bronze. Les SNPs sélectionnés à cette époque calibrent des réponses immunitaires forte mais équilibrés vis-à-vis de l’immunité Th1 (contre les pathogènes intracellulaires) et Th2 (contre les pathogènes extracellulaires et les parasites). L’hygiène de notre époque a plus fortement décru les besoins en immunité Th2 qu’en immunité Th1, ce qui fait écho au déséquilibre Th1/Th2 observé dans la SEP. Ceci ouvre une piste pour faire le lien entre l’augmentation actuelle de l’incidence de la SEP et les changements d’environnement et de mode de vie de notre époque.
Références
Barrie, W., Yang, Y., Irving-Pease, E.K., Attfield, K.E., Scorrano, G., Jensen, L.T., Armen, A.P., Dimopoulos, E.A., Stern, A., Refoyo-Martinez, A., Pearson, A., Ramsøe, A., Gaunitz, C., Demeter, F., Jørkov, M.L.S., Møller, S.B., Springborg, B., Klassen, L., Hyldgård, I.M., Wickmann, N., Vinner, L., Korneliussen, T.S., Allentoft, M.E., Sikora, M., Kristiansen, K., Rodriguez, S., Nielsen, R., Iversen, A.K.N., Lawson, D.J., Fugger, L., Willerslev, E., 2024. Elevated genetic risk for multiple sclerosis emerged in steppe pastoralist populations. Nature 625, 321–328. https://doi.org/10.1038/s41586-023-06618-z
Bjornevik, K., Cortese, M., Healy, B.C., Kuhle, J., Mina, M.J., Leng, Y., Elledge, S.J., Niebuhr, D.W., Scher, A.I., Munger, K.L., Ascherio, A., 2022. Longitudinal analysis reveals high prevalence of Epstein-Barr virus associated with multiple sclerosis. Science 375, 296–301. https://doi.org/10.1126/science.abj8222
Froment, A., 2013. Anatomie impertinente: Le corps humain et l’évolution. Odile Jacob, Paris.
International Multiple Sclerosis Genetics Consortium, 2019. Multiple sclerosis genomic map implicates peripheral immune cells and microglia in susceptibility. Science 365. https://doi.org/10.1126/science.aav7188